شبیه سازی جداسازی DNA با کامسول
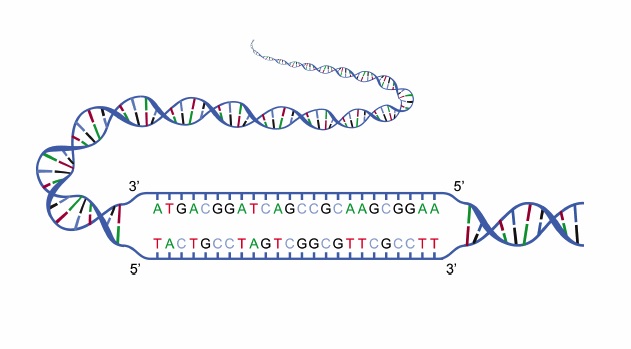
شبیه سازی جداسازی DNA با کامسول نگاهی دقیق تر به آرایش ژنتیکی ما ساختار مولکولی DNA پیچیده است: این یک پلیمر دو مارپیچ است که از یک زنجیره طولانی از نوکلئوتیدها تشکیل شده است. مطالعه DNA با تجزیه نمونه به قطعات با اندازه های مختلف بسیار آسان تر است.
جفت های نوکلئوتیدی یا باز DNA عبارتند از گوانین (G)، آدنین (A)، تیمین (T) و سیتوزین (C). محققان سعی میکنند توالیهای این «حروف» ژنتیکی را در زمینههایی مانند توالییابی ژنوم و تشخیص پزشکی معنا کنند، برای مثال، ژنها را پیدا کنند و ببینند چگونه با هم در یک ارگانیسم کار میکنند. انجام این کار بدون جداسازی DNA آسان نخواهد بود – به هر حال، ژنوم انسان دارای بیش از 3 میلیارد جفت باز DNA است! نمونه های دیگری از تجزیه و تحلیل DNA اخیراً مورد توجه قرار گرفته اند. ممکن است با کیت های تست DNA پستی آشنا باشید که به شما کمک می کند درباره اصل و نسب خود اطلاعات بیشتری کسب کنید. هنگامی که یک شرکت آزمایش ژنتیک نمونه DNA شما را دیجیتالی می کند، مانند یک رشته طولانی از نوکلئوتیدهای G، A، T و C به نظر می رسد. این شرکت ها از الگوریتم هایی برای مقایسه قطعه DNA شما از ژنوم با مجموعه داده های مرجع استفاده می کنند. سپس، الگوریتم تعیین میکند که نمونه DNA شما چقدر با هر مجموعه مرجع مطابقت دارد تا ببیند به احتمال زیاد به کدام گروههای اجدادی تعلق دارید. الگوریتم فقط به اندازه مجموعه های مرجع آن خوب است، بنابراین برخی از گروه های اجدادی ممکن است در مقایسه با دیگران در پایگاه داده کمتر ارائه شوند.
در زمینه هایی مانند پزشکی قانونی، پروفایل DNA به دانشمندان کمک می کند تا نمونه هایی از مواد ژنتیکی را با هم مقایسه کنند. از آنجایی که نادر است که دو نفر الگوی DNA یکسانی داشته باشند، دانشمندان پزشکی قانونی می توانند الگوهای موجود در برش های مولکول های DNA را با پایگاه های داده مرجع، مانند سیستم فهرست ترکیبی DNA (CODIS) که توسط FBI ایالات متحده مدیریت می شود، مقایسه کنند. با این حال، سیستمهایی مانند CODIS محدود به پروفایلهای DNA هستند. محققان شروع به استفاده از پایگاههای اطلاعاتی اجداد مانند موارد ذکر شده در بالا برای گسترش جستجوهای خود از طریق مفهومی به نام DNA خانوادگی کردهاند. به عنوان مثال، در سال 2018، پلیس در حال بررسی پرونده قاتل گلدن استیت، DNA صحنه جنایت را در پایگاه داده سایت شجره نامه بررسی کرد و یک تطابق جزئی با یکی از بستگان دور پیدا کرد. در نهایت، این به آنها کمک کرد تا جستجوی خود را محدود کرده و مظنون را شناسایی کنند. برای آموزش کامسول همین الان اقدام کنید.
بررسی پیوندهای تکه تکه شده در یک زنجیره نوکلئوتیدی با جداسازی DNA
یکی از روشهای رایج برای جداسازی مولکولهای DNA – عمدتاً در پزشکی قانونی – الکتروفورز ژل است که شامل مهاجرت مولکولهای اسید نوکلئیک با بار منفی از طریق یک ژل است. هنگامی که جریان الکتریکی اعمال می شود، مولکول های کوچکتر سریعتر از مولکول های بزرگتر در ژل حرکت می کنند، بنابراین قطعات بر اساس اندازه به نوارهایی از هم جدا می شوند. برای تجسم این جداسازی، از رنگ رادیواکتیو استفاده میشود. محققان دانشگاه علم و فناوری میسوری برای اینکه بفهمند آیا آنها میتوانند در زمان با یک نرمافزار شبیهسازی تجاری در دسترس صرفهجویی کنند، سیستم تله آنتروپیک و شبیهسازی دینامیک پلیمری خود را با استفاده از COMSOL Multiphysics راهاندازی کردند و مقایسه کردند. نتایج آنها به داده های تجربی است.
آموزش comsol شبیه سازی جداسازی DNA با کامسول، تیم تحقیقاتی متشکل از جونتاک پارک، جیمز جونز، میامای پالانیاپان، سامان منجزی و بهروز بهدانی میگویند که «دینامیک DNA در شبیهسازی میکروکانال چالش برانگیز است، زیرا دو شبیهسازی مختلف – محاسبه میدانی در هندسه میکروسیالات پیچیده و دینامیک مولکولهای پلیمری – باید ترکیب شود.» خوشبختانه، آنها اضافه می کنند، “COMSOL® می تواند نسبتاً به راحتی شبیه سازی قبلی را انجام دهد” و “COMSOL می تواند صفحه جدیدی را در ناحیه شبیه سازی DNA یا مولکول تک پلیمری باز کند.”
با استفاده از افزودنی ماژول ردیابی ذرات، تیم شبیه سازی دینامیک براونی زنجیره DNA را انجام داد. این زنجیر بهعنوان یک مدل تک پلیمری و زنجیرهای در یک سیال نیوتنی با کمک ماژول CFD راهاندازی شد. در مورد خود مهرهها، آنها به عنوان ذرات براونی در نظر گرفته میشوند تا حرکات تصادفی زنجیره را هنگام حرکت در حلال اطراف محاسبه کنند. مطالعه مقاله قبلی در مورد شبیه سازی پورت دایره ای در ماژول رادیویی RF کامسول را از اینجا بخوانید.
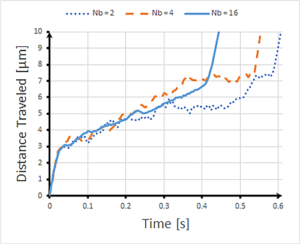
برای توصیف نیروی فنر بین هر مهره، آنها از مدل معروف دیگری به نام زنجیره کرم مانند (WLC) استفاده کردند که رفتار پلیمرهای نیمه انعطاف پذیر را توصیف می کند. در کنار WLC، تیم تحقیقاتی از پتانسیل لنارد-جونز برای جلوگیری از نفوذ مهرهها به یکدیگر استفاده کردند. سپس، محققان مسیر مرکز جرم مولکولهای DNA را بر اساس طول، در Nb = 2، 4 و شبیهسازی کردند. 16 طول مهره، به عنوان آنها به طور دوره ای به کانال منقبض جریان. مسیر حرکت برای هر یک از مولکول هایی که در یک فاصله حرکت می کنند در زیر نشان داده شده است. همانطور که از بردار میدان الکتریکی بالا انتظار میرود، مولکولها در کانالهای باریک سریعتر حرکت میکنند، و هر چه مولکول طولانیتر باشد (هرچه دانههای بیشتری داشته باشد)، سریعتر حرکت میکند. در همین حال، مولکولهای کوتاهتر، در طول مسیر خود دارای سرعت کاهشیافتهای هستند و توزیع مولکولهای DNA نشاندهنده الگوی انتشار بیشتری است که با دور کردن آنها از مناطقی که میدان الکتریکی قویترین است، سرعت کلی را در کانالها کاهش میدهد.